「サイエンティフィックPythonのための」IDE、Spyder上にてScientific Python Lecturesの実習中。前回は定番の画像のフィルタをいつくか適用した結果を並べて悦にいりました。今回は画像のモフォロジー処理です。画素の集合を対象にその構造や形状を操作、解析するための処理です。
※「 ソフトな忘却力」投稿順 Index はこちら
※Scientific Python Lectures様のコースは例題だけでなく、エクササイズなども充実、それを全部順番に解いていったら必ずや立派な人になれるだろ~と思います。でも老い先短い年寄には量が多過ぎて多分死ぬまでに終わりません。適当な練習でお茶を濁してます。今回の「お勉強」は「5.10 Image manipulation: scipy.ndimage」の「5.10.3 Mathematical morphology」です。
モフォロジー処理
2値化画像のモフォロジー処理については、以下の別シリーズ過去回で練習してます。
手習ひデジタル信号処理(140) Scilab、{IPCV}、モルフォロジー処理
今回は、上記の過去回でやった以下の処理を、SciPyでも実行できることを確かめたいと思います。
-
- opening
- closing
- hit_or_miss
- gradient
処理の練習台にしたのは以下の画像です。黒字に白で稚拙な「う」という文字を描いたものです。
モフォロジー処理のサンプルスクリプト
以下では、上記の画像について4種のモフォロジー処理をかけてます。しかし、出だしでちょっと躓きました。Windowsのペイントを使って「2値のBMP」画像(256x256サイズ)として上記画像をセーブしたのです。確かに2値になっているみたいなのですが、matplotlibのimread関数で読み込んだ中身を見ると、単純な2次元配列ではなく、ピクセル方向に4要素ある3次元の配列にみえます。値は2値っちゃ2値ですが、今回のモフォロジー処理は、グレースケールやバイナリを当てにしている関数どもだったりするので、変換必須。しかし、調べてみたら、SciPyにはバイナリ画像に変換する関数が無いみたい。仕方がないので、Googleの生成AI、Gemini 2.5 Flash様にそれをするための方法をお聞きして以下のコードの先頭部分に仕込みました。なんだかな~、AIに頼り切っているのう。
#!/usr/bin/env python3
# -*- coding: utf-8 -*-
"""
Created on Thu Jul 10 12:57:44 2025
@author: jhalfmoon
Morphological Operations sample
"""
import numpy as np
import matplotlib.pyplot as plt
import scipy as sp
def plotFig(fig, title):
plt.imshow(fig, cmap="gray")
plt.title(title)
def main():
img = plt.imread('sampleFigMono.bmp')
imgGrey = np.mean(img, axis=2)
threshold = 127
binary_array = (imgGrey > threshold).astype(int)
imgOpening = sp.ndimage.grey_opening(binary_array, size=(8,8))
imgClosing = sp.ndimage.grey_closing(binary_array, size=(7,7))
strc = np.ones((5,5))
imgHitMiss = sp.ndimage.binary_hit_or_miss(binary_array, structure1=strc)
imgGrad = sp.ndimage.morphological_gradient(binary_array, size=(3,3))
plt.figure(figsize=[12.8,4.8])
plt.subplot(151)
plotFig(binary_array, "Original(binary)")
plt.subplot(152)
plotFig(imgOpening, "Opening 8x8")
plt.subplot(153)
plotFig(imgClosing, "closing 7x7")
plt.subplot(154)
plotFig(imgHitMiss, "HitMiss")
plt.subplot(155)
plotFig(imgGrad, "Gradient")
plt.tight_layout()
plt.show()
if __name__ == '__main__':
main()
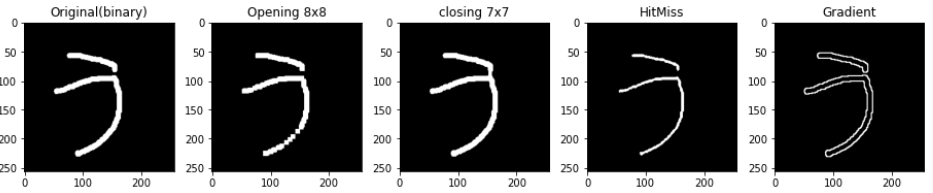
スクリプトの実行結果
一番左が(2値変換後の)オリジナル画像です。その右隣りが、8x8ピクセルサイズのオプニンク処理を行ったもの。例えていうと8x8サイズの棒を作ってオリジナル画像の穴の開いているところ(白)にグリグリと突っ込んで「通せる」ところだけをとりだしたようなもんです。ホントか?一方そのお隣はクロージング処理、こちらは7x7サイズ。すると「隙間」が埋まってして繋がってしまうのを目撃。お次のHitMiss処理するとなんだか全体が細くなっている気がするし、一番右のGradient処理では端っこ部分のみが白抜きになり二重線ぽくなってます。
一応、モルフォロジーできてるみたい。ホントか?