前回は全粒粉に含まれる微量な銅の分析でした。今回も「分析」ネタがつづきます。同じ資料について異なる研究室(研究所?)で分析を行って、その結果を比較してみたものみたいです。研究室毎のバイアス?だけでなく資料と研究室の「交互作用(Interaction Effect)」の有無に関心があるみたい。まあいろいろあるのね。
※「データのお砂場」投稿順Indexはこちら
Co-operative Trial in Analytical Chemistry
今回のサンプル・データ・セットについての解説ページが以下に。
https://stat.ethz.ch/R-manual/R-devel/library/MASS/html/coop.html
7つの検体が、3つのバッチに分けられて、6つの研究室(研究所?)に送られたみたいです。それぞれの研究室で対象物質(上記の解説記事では結果の単位のみ記載されており、物質名には言及なし。漏れ伝わるところによると分析しているのはビタミンCらしいっす。知らんけど)の含有用が測定され、比較された、と。
当然のごとく、研究室毎のクセ、ここは高めに出るとか、低めな結果を出すとかの傾向があり。さらにいうと、全ての検体で同じようなクセが出るわけでもなく、検体と研究室の組み合わせでも「交互作用」というものを検出したいみたいです。サンプルデータベースとしては、
二元配置の分散分析(ANOVA)の練習用
であるようです。研究室(L1からL6)、検体(S1からS7)、バッチ(B1からB3)についてはカテゴリカル変数です。なお、バッチというのは検体の処理のためにまとめた単位だと思うのですが、そいつがどのような影響を与えているのかは定かではありませぬ。同じ検体が異なるバッチに入っている、と思われるので、結果がブレブレだとかが分かるの?どうなんだろ。
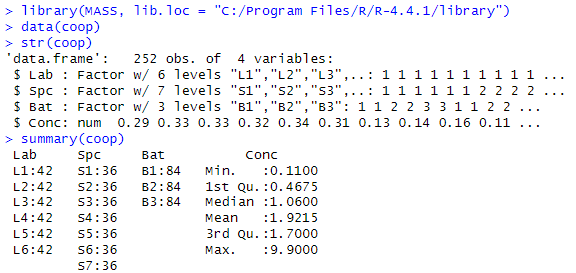
まずは生データ
上記を拝見するに、各研究室ではそれぞれ42回の測定を行っているみたい。検体は7種類、バッチは3種類なので、7x3x2=42ということなのかな? バッチの中に同じ検体が2回入っている見当。
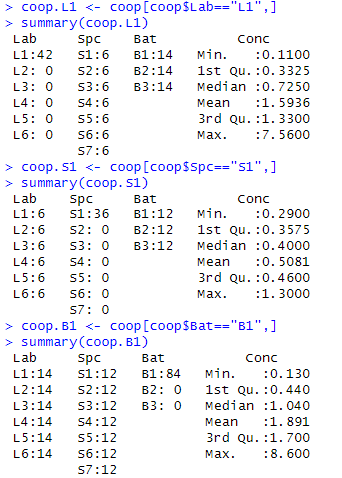
以下のようにして「サブセット」を取り出して確認してみます。
coop.L1 <- coop[coop$Lab=="L1",] summary(coop.L1) coop.S1 <- coop[coop$Spc=="S1",] summary(coop.S1) coop.B1 <- coop[coop$Bat=="B1",] summary(coop.B1)
まあ、お惚け老人の理解とは矛盾していないみたい。大丈夫か?
何がなくともビジュアライゼーション
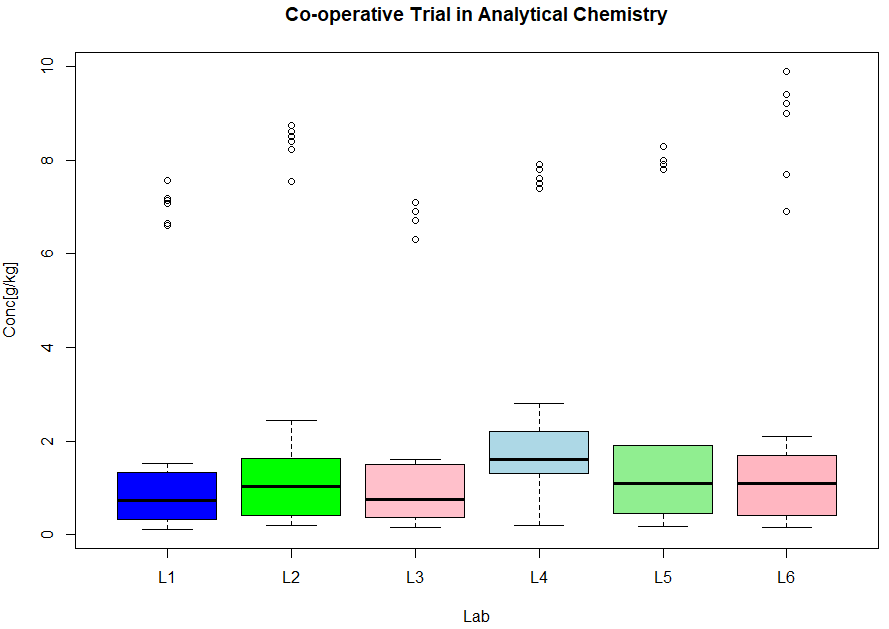
まずは、各カテゴリカル変数毎に分類した箱ヒゲ図を描いて、雰囲気を味わってみます。まずは研究室毎。
labcol <- c("blue", "green", "pink","lightblue", "lightgreen", "lightpink")
boxplot(Conc ~ Lab, data = coop, main = "Co-operative Trial in Analytical Chemistry", xlab = "Lab", ylab = "Conc[g/kg]",col = labcol)
それなりにバラついているのね、中でもL4の研究室、ちょいと高めに出過ぎてないかい? 知らんけど。
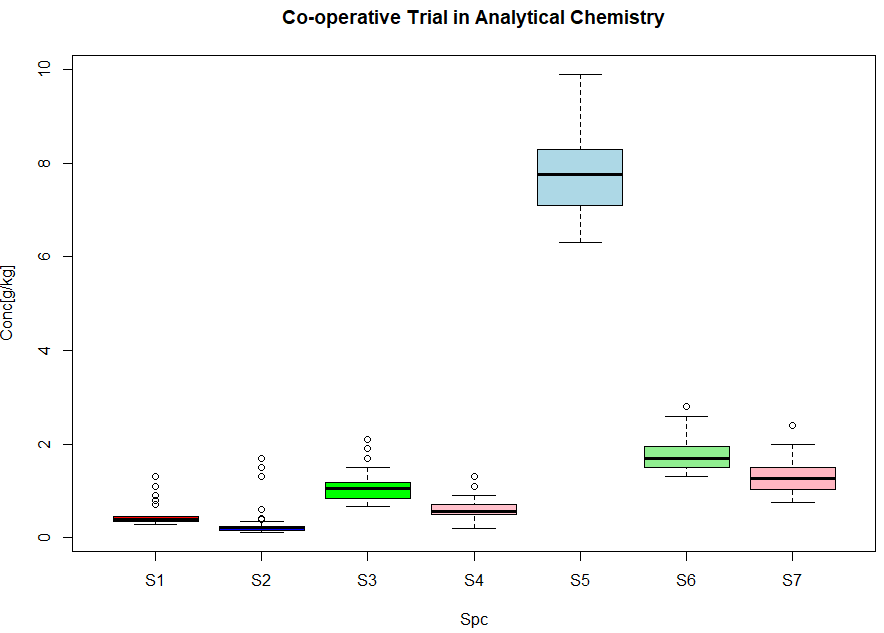
つづいて標本別でのプロットの指示が以下に。
spccol <- c("red", "blue", "green", "pink","lightblue", "lightgreen", "lightpink")
boxplot(Conc ~ Spc, data = coop, main = "Co-operative Trial in Analytical Chemistry", xlab = "Spc", ylab = "Conc[g/kg]",col = spccol)
こちらは、S5という標本だけが「濃ゆい」みたい。これは意図的に濃くしたのかね? S1とS2、S3とS4、S6とS7は「似たような」感じだと。
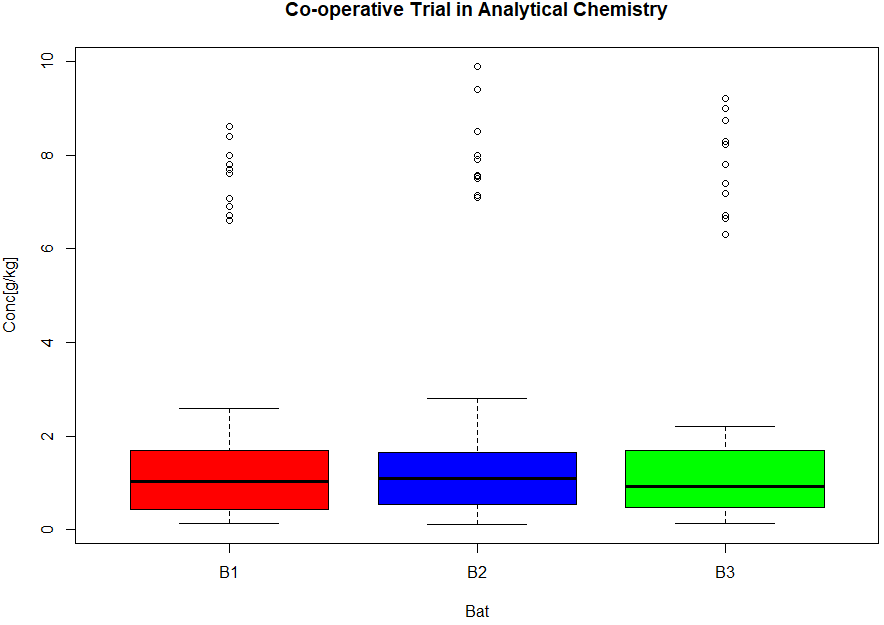
さて、一応「バッチ」についても見てみましたぞ。
batcol <- c("red", "blue", "green")
boxplot(Conc ~ Bat, data = coop, main = "Co-operative Trial in Analytical Chemistry", xlab = "Bat", ylab = "Conc[g/kg]",col = batcol)
お惚け老人の期待どおりか、3バッチについては「似たような」箱ヒゲ図が描けました。でも、よく見ると多少のバラツキはあり。まったく同じにはならんね。
研究室と標本の「交互作用」
以下のプロットにて「交互作用」の雰囲気を確かめてみます。
interaction.plot(coop$Spc, coop$Lab, coop$Conc, col = 1:6, lty = 1, lwd = 2, trace.label = "Lab", xlab = "Spc", ylab = "Conc[g/kg]", main = "Co-operative Trial in Analytical Chemistry")
最初にカテゴリカル変数2個と数値変数1個が列挙されとりますが、以下のような順番です。
-
- x.factor横軸(X軸)に並べる因子
- trace.factor折れ線グラフの「線」として区別する因子
- response縦軸(Y軸)にくる数値データ
なおデフォルトでは、Y軸の数値データは「平均値」が計算されるみたいです。結果プロットが以下に。黄色の丸はお惚け老人がマークしたところ。
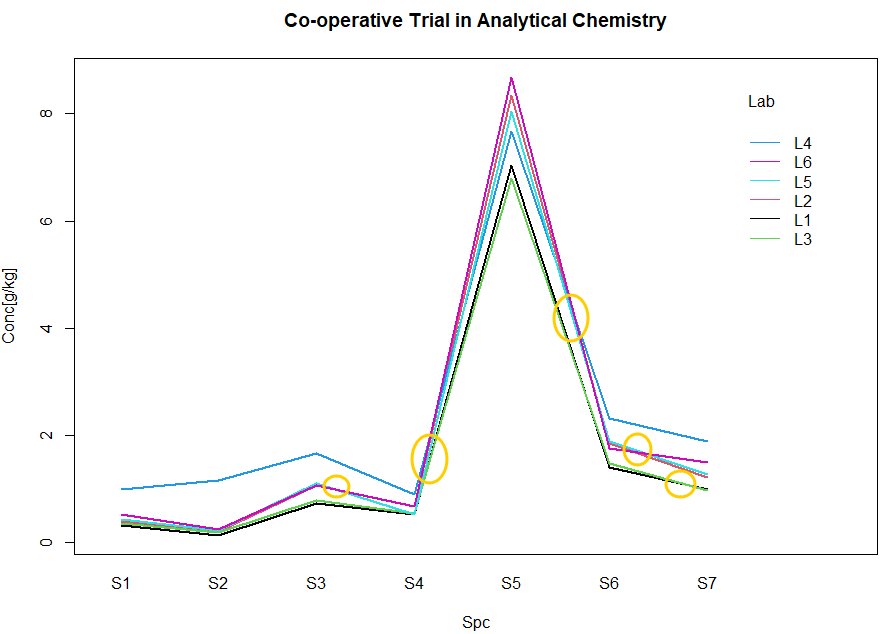 たとえば、青のL4研究室は「いつも上目の結果」を出しがちに見えますが、こと「濃ゆい」S5の検体に関しては他の研究室の方が「上」にきてます。似たような「順位の逆転」は黄色のマル印のところにあり(拡大したらもっとあるかもだけれども。)
たとえば、青のL4研究室は「いつも上目の結果」を出しがちに見えますが、こと「濃ゆい」S5の検体に関しては他の研究室の方が「上」にきてます。似たような「順位の逆転」は黄色のマル印のところにあり(拡大したらもっとあるかもだけれども。)
Two-way ANOVA(二元配置分散分析)
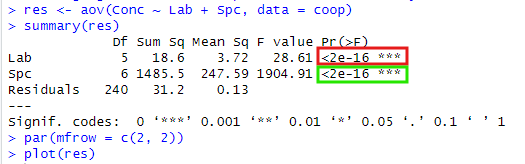
以下のようにして二元配置分散分析を実施してみましたぞ。
res <- aov(Conc ~ Lab + Spc, data = coop) summary(res) par(mfrow = c(2, 2)) plot(res)
p値をみると、Labに関してもSpcに関してもバッチリ「有意」みたいっす。
上記をみても、忘却力の老人はほぼほぼグラフの見方を忘れとります。以下のような過去回で「分散分析」繰り返した筈なんだが。
データのお砂場(14) R言語、InsectSprays、殺虫剤の効きの分散分析とな?
データのお砂場(21) R言語、OrchardSprays、果樹園用農薬?の効果とな?
データのお砂場(22) R言語、PlantGrowth、植物の成長実験、無味乾燥?
データのお砂場(48) R言語、chickwts、ニワトリさんの体重、「再び」とな
データのお砂場(64) R言語、morley、実際はMichelson、光速の測定
データのお砂場(68) R言語、npk、古典的なn,p,k要因実験とな。NPKって何?
データのお砂場(88) R言語、warpbreaks、織機での「糸切れ」回数、糸と張力
ぼーっとして「繰り返して」いても身につかんということか。サッパリだな自分。