R言語のサンプルデータセットをABC順(大文字先)で端から試してみていますが、お薬ネタ?も時々ありますな。今回は、ピューロマイシンという抗生物質だそうです。この抗生剤を使ったときと使わぬときでの何やら反応速度の違いを測定したデータみたい。これまた分かったような分からぬような。でも処理の方法は以前の回同様で良さそう。
※「データのお砂場」投稿順Indexはこちら
データセットの解説ページへのリンクは以下です。
Reaction Velocity of an Enzymatic Reaction
データに関する多少の説明、conc(濃度)はppm だとか、rate(反応速度)の単位は count毎分毎分(反応すると得られる放射性物質の毎分カウントの値を初期状態からの変化としてとらえているみたい)とか、多少の説明はあります。しかし、当然ながらピューロマイシンって何といった説明はないです。データの出典は示されているので、知りたければソッチを当たってくれという感じ?まあ、統計処理のサンプルなので当然ですが。
WebでPuromycinを検索すると、お薬として結構使われているみたいでいろいろヒットしてきます。お薬ネタの深みにハマらぬよう今回は通りすぎます。
また、お薬ネタで似たような処理を行っている以前の記事は以下です。
データのお砂場(13) R言語、Indometh、インドメタシンの薬物動態とな?
今回は上記記事で行っている処理を「ブラッシュアップ」? ホントか?
生データの様子
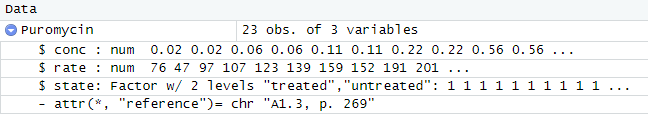
サンプルデータを選択して、データ形式を調べ、先頭データを眺めるという方法でいつも確認しています。こんな感じ。
でも実際は、Rstudioを使わせていただいているので、class()でデータに触った瞬間、以下のように変数ウインドウに内容がネタバレしております。実際はhead()しないでもどんなデータか分かっておるわけで、すみません。
勝手グラフ化
処理例では普通のplotを使って地味にグラフを描いてました。こちらでは以前の回に習い、ggplot2を使って少し見栄えのする?グラフとしてみます(ggplot2はもっと華やかなグラフ表現もいろいろできます。以下で使っているのはデフォルトのまま。)
処理の命令が以下に
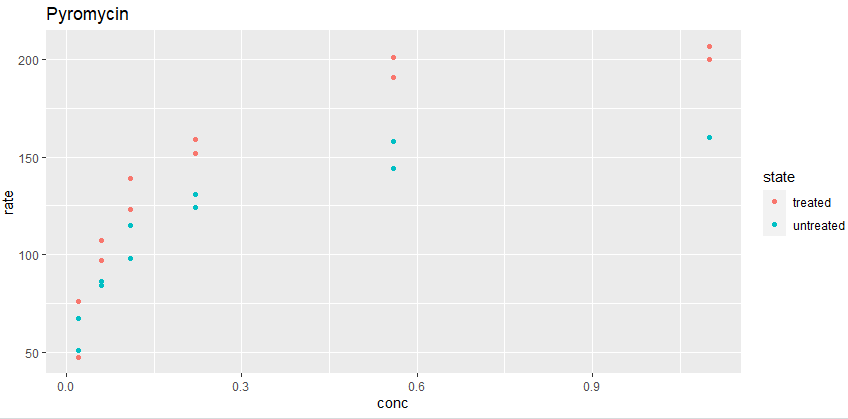
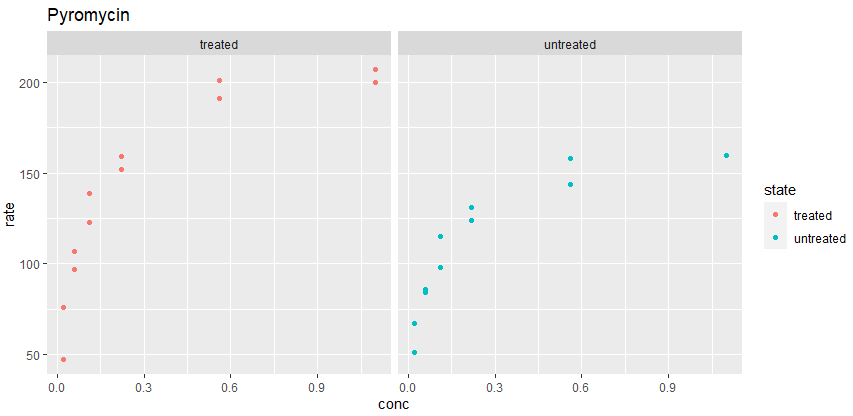
g0<-ggplot(data=Puromycin, aes(conc, rate, color=state))+geom_point()+labs(title="Pyromycin") g1<-g0 + facet_wrap(vars(state))
g0を表示させると以下のようです。ピューロマイシンで処理した(treated)方のオレンジが、処理しなかった(untreated)方の緑より明らかにレートが高いです。
g1のファセットを切ったグラフがこちら。今回は treatedとuntreatedの2つだけの比較なので、わざわざファセットとるよりは、上記のようなグラフの方が良いように思われます。
例題処理例
処理例に書かれていた地味なグラフの生成コマンドが以下に。
グラフ表現は地味ですが、ラベルが充実しているので、情報量が多くてこちらはプロ仕様?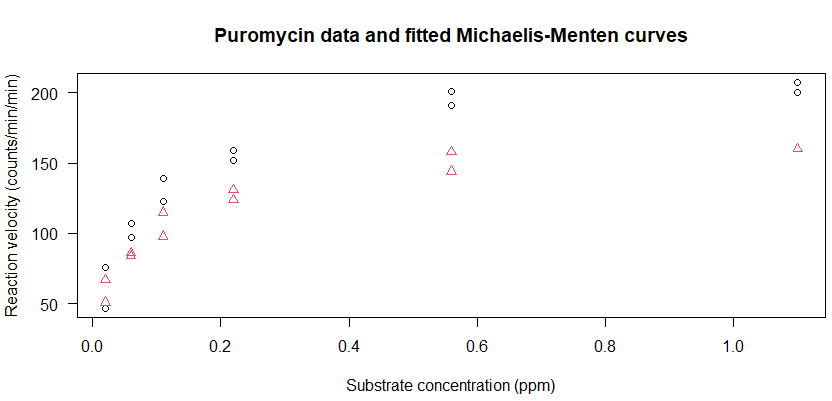
上記のグラフが出て来たので「回帰曲線」を引かずにはいられませぬ。ここでは、nls()関数を使って、以下のような関係に回帰させてます。
rate ~ vm * conc/(K + conc)
なぜ、このような式を思いついた(思いつきじゃないだろ~)のかは特に説明はありません。私は分かりません。多分、この式でうまく説明つくのでしょう、としか言いようがないです。
nls()つかってvmとKを推定しているところが以下に。fm1の方が treated なデータ、fm2の方が untreated なデータの処理です。
わけわかってませんが、上記で vm と K の値が決まったので、それを回帰曲線に当てはめて描いてみることが可能。その処理例が以下に。
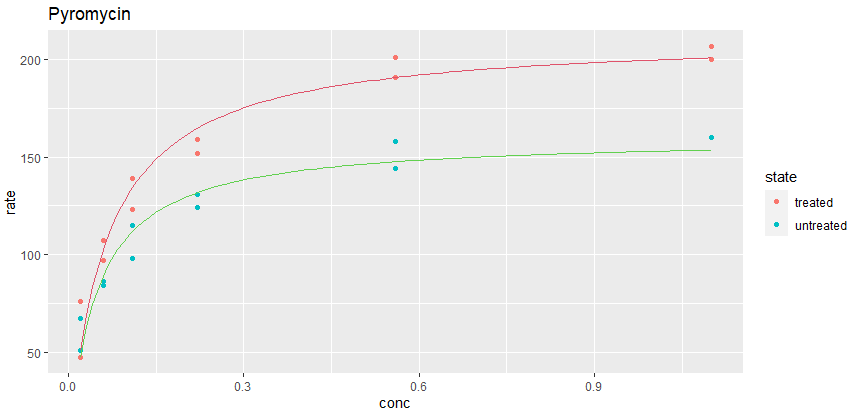
上記処理により、曲線と凡例がグラフに追加されました。処理例、いい感じだな。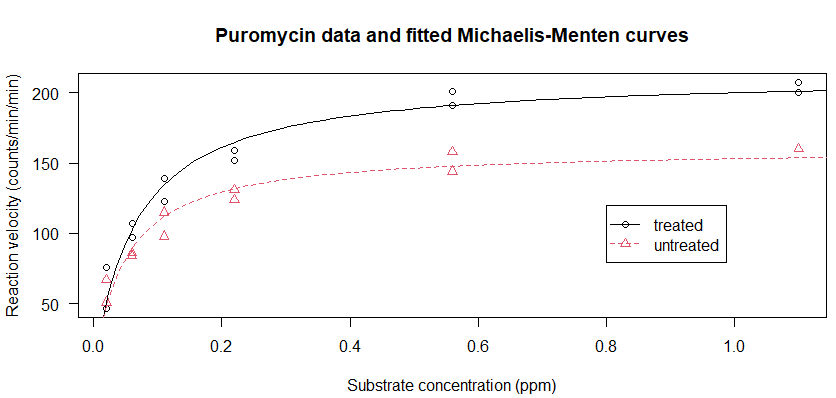
勝手グラフ側にも回帰曲線追加したい
上記のグラフで満足していると、処理例をそのまま実施しただけで、こちらの進歩はまったくないです。先にやったggplot2のグラフに、回帰曲線を追加してみることにしました。すでにnlsでvmとKという定数の推定値が求まっているので、処理例で使っていた式をそのまま当てはめて線をグラフに上書きしてみます。こんな処理シーケンス。
g20<-ggplot(data=Puromycin, aes(x=conc, y=rate, color=state))+geom_point()+labs(title="Pyromycin")
g21<-g20 + stat_function(fun=function(x){coef(fm1)[[1]] * x/(coef(fm1)[[2]] + x)}, color=2)
g22<-g21 + stat_function(fun=function(x){coef(fm2)[[1]] * x/(coef(fm2)[[2]] + x)}, color=3)
g22
上記の処理シーケンスで描きなおした ggplot2 のグラフ(冒頭のアイキャッチ画像)がこちら。勿論、処理例のグラフと同じものですが、見た目がちょいと違います。XYのラベル追加しておいた方がよかったな。いまや遅い。
お薬ネタ、だんだん慣れてきた?