前回、美麗なグラフが描けるようになって喜びましたが、今回は「またしても」なんだか分からないデータに遭遇です。ELISAアッセイ?人工無能なイライザ様なら遥か昔にお付き合いしたことありますが、皆目見当もつきません。抗原とか抗体を酵素反応を利用して調べるためのものみたい。それをまたしても非線形回帰分析せよ、と。
※「データのお砂場」投稿順Indexはこちら
R言語に漏れなくついてくる(多分?)サンプルデータセットをABC順に端から開いてみております。開くたびに皆目見当のつかない世界が広がっていて目が回ります。その上、R言語素人、統計も素人で、まさに3重苦。しかし、新世界を覗き見るべく本日もデータに戯れたいと思います。ヤッツケでご乱心。
DNaseデータセット
まずは、データセットについて解説しているページへのリンクを貼っておきます。
この「説明」を見た時、機械工学科卒、その後マイコン屋の私メは、まったく分からんでしした。しかし、今やネットのお陰で分かったような気には簡単になれると(分かってなくても。)
まずElisa assayについては、以下のページが分かり易いと思いました(個人の感想です。)
上記から1か所引用させていただきます。
試料溶液中に含まれる目的の抗原あるいは抗体を、特異抗体あるいは抗原で捕捉するとともに、酵素反応を利用して検出・定量する方法です。
勝手な解釈だと、「目的の物質の量が多いと色が濃くなる」みたいです。それで縦軸に吸光度、横軸に抗原量をとるみたい。そしてDNaseですが
デオキシリボヌクレアーゼ
という「酵素群の総称」らしいです。お名前から想像できるようにDNAにも関係しているみたい。
まあ、何となく、ホンワカしたレベルで先に進んでいきませう。
まずはデータを吟味
何回かやって、ここの手順はほぼ固まりましたな。まずはデータセットをロードして、そのデータセットがどのような形式のデータかを調べ、そして「とりあえず」生データをダラダラ出力させて眺めてみる、と。こんな感じ。
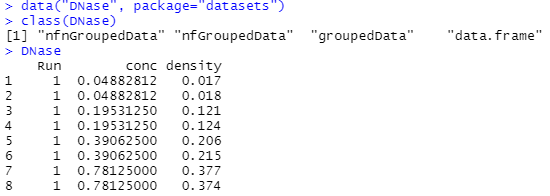
上記は8点で切っていますが、実際には176個のデータ点が含まれます。例によって遅延評価オブジェクトであり、nfnGroupedData以下良く分からない「デコレーション」を含みますが、ここまで学んできたとおり単なる「data.frame」として扱ってしまいます。
ggplot2でグラフ化
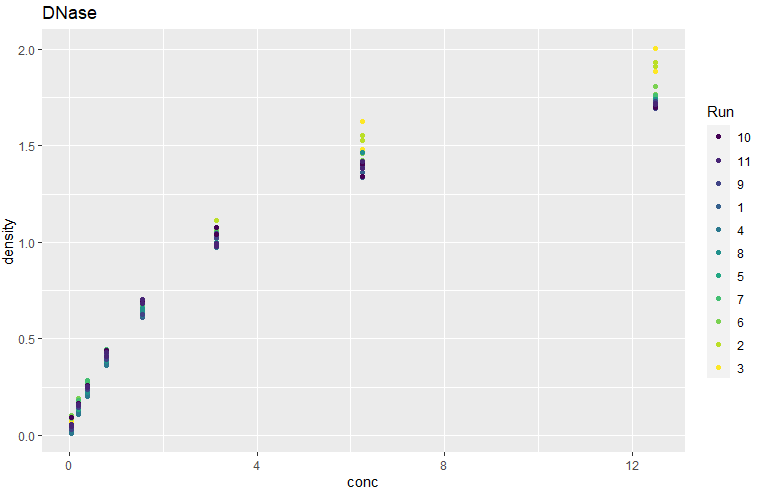
前回、ようやく ggplot2に目覚めたので、さっそくデータ全体をグラフ化してみます。x軸がconc、既知のタンパク質の濃度、y軸がdensity、測定した光学的な「密度」(単位は無し)です。これをプロットさせるコマンドが以下に。
![]() プロットの結果を冒頭のアイキャッチ画像に掲げましたでござりまする。前回の鳥さんデータのバラツキ具合に比べると、結構まとまった感じがします。
プロットの結果を冒頭のアイキャッチ画像に掲げましたでござりまする。前回の鳥さんデータのバラツキ具合に比べると、結構まとまった感じがします。
上記は「素のデータ」のままグラフ化しましたが、どうもこのデータの解析にあたっては、X軸を対数でとるべきなようです。x軸を対数にして描きなおすためのコマンドがこちら。
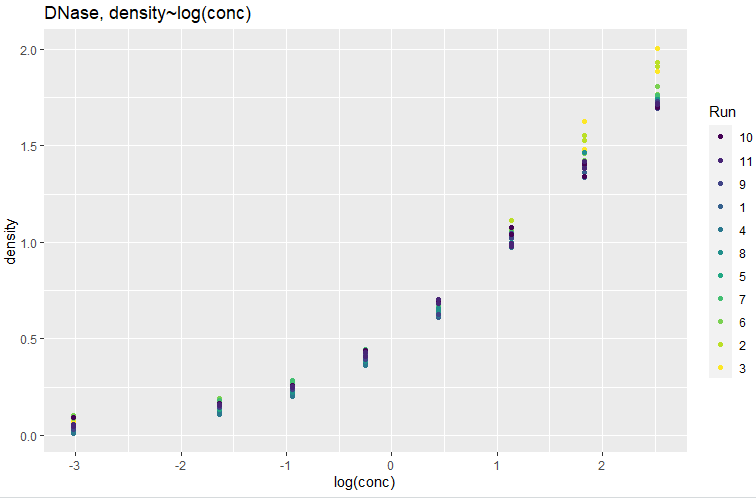
上のプロットを見て、MOSFETの特性グラフを想像してしまいました。実は似たようなもん、だと思うと気持ちの敷居が下がります(嘘です。)
非線形回帰分析をせよ、と
解説ページには、処理例が示されています。そこを見ると、nls()で解析するようになってました。再び 非線形回帰分析 の登場であります。まあ、ABC順に端からやってきているので、ムツカシイのが混ざっているのはショウガナイですが、またですか。
このnls()を使うためには、関数のモデルというかを指定しないとならず、そこで使われている関数が何やら恐ろし気です。しかし、とても良いページを見つけました。
どうもSelf-startingというのは、初期値を明示的に与えなくてもよきようにやってくれるものみたいです(ホントか?)そして上記のページにはそれぞれの関数のグラフとAsymとかxmid、scalといったパラメータが何なのか式とグラフで示されておりますのじゃ。グラフを見ると「分かった気」になり(ホントのところは不明だが)、「恐れ」が和らぐのを感じまする。
さて、第1のモデルがこちら。
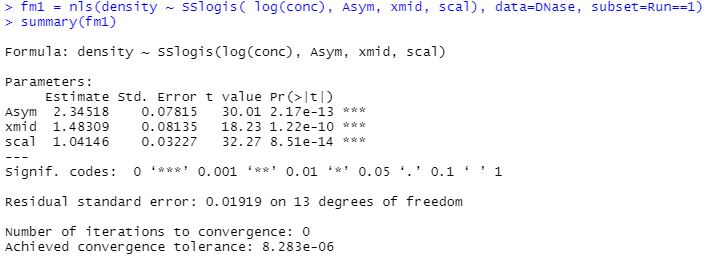
ホントのところの意味は不明であるものの、上記のページのグラフを参照すれば、こんな感じのグラフに当てはめてみているのね、と納得?
そして第2のモデルがこちら。関数のグラフを見れば、第1のモデルと「似ている」けれどもちょっと「自由な感じ」がいたします。
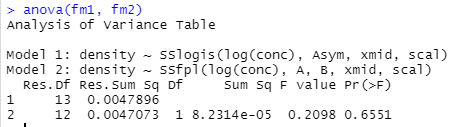
分散分析
をせよとの思し召しでした。これはダメだ。改めて勉強しないと。今回は理解は速攻でパス。しかし、実行するのは簡単なので、やってみましたぞ, anova()。