サンプルデータセットを眺めていると未知の世界が突然開けることがあります。今回のbootパッケージのcavデータセットもそのようなもののひとつです。皆さん「漏れなく持っているけど」その存在を知っているのは「関係者のみ」?「その関係先」にはコンドロイチンとかヒアルロン酸とか聞いた名前もチラホラ登場。このデータをどうしろと?
※「データのお砂場」投稿順Indexはこちら
※ R言語向けのパッケージ boot には、サンプルデータセット多数が含まれております。それを ABC順に「舐めて」ます。今回のサンプルデータセットはcavとな。
Caveolae(カベオラ)
Wikipediaによると「カベオラは1955年に山田英智によって発見された」とあります。日本人発見だったのね。山田先生の1955年の以下の論文へのリンクも張られています。
Yamada, E. (1955-09-25). “The fine structure of the gall bladder epithelium of the mouse”. The Journal of Biophysical and Biochemical Cytology 1 (5): 445–458. doi:10.1083/jcb.1.5.445. ISSN 0095-9901. PMC 2229656. PMID 13263332.
上記のリンクは、National Library of Medicineというサイトへのリンクでそこで1955年の論文のアブストラクトを読むことができます。ありがたいことです。そこからcaveolaeという言葉が使われている1文を引用させていただきます。
Very small pit-like depressions, representing caveolae intracellulares, are encountered along the cell membrane of the microvilli.
『Very small pit-like depressions』だそうな。サイズ的には50nmくらいらしいデス。チイセーです。どうも細胞膜上に点々と存在する「窪み」らしいです。現物写真が見てみてーということで探したら以下のところに写真が見つかりました。沖縄科学技術大学院大学様
上記のサイトにバッチリ?な写真があります。また、以下の東京大学のページには、カペオラの模式図があります。
細胞内取り込みのメカニズムを解明 分子モータータンパク質KIF13Bの役割
このカベオラという洞窟(穴ぼこ)は、なんやら細胞膜の物質取り込みに関わるものみたいっす。知らんけど。
その辺、素人にも分かり易く?説明してくださっていたのが、Glycoforum様の以下のページです。
上記のページを読ませていただくと、ちょっとは分かったような気になります。素人の錯覚?
さて、上記ページのGlycoforumという組織って何?と調べてみたら、生化学工業株式会社様がお金を出して運営されている「ジャーナル」でした。その趣旨を説明する一文を引用させていただきます。
当社は、学問尊重の理念のもと、糖質科学の発展の一翼を担う企業としての責務、そして糖質科学への恩返しの旨趣を込めて、純粋に学術的な内容のオープンアクセスジャーナルとしてGlycoforum®を運営しています。
おおそうだったのね。なお、生化学工業株式会社は、1950年に「コンドロイチン硫酸」を工業化した会社で、他の主力製品としては「ヒアルロン酸」があるようです。あちこち(主に年寄の立ち回り先)で目にすることが多いコンドロイチンとかヒアルロン酸ですが、その根っこに位置する(でも表には出てこない)会社なのね。。。
さてカペオラの発見者の東大名誉教授、山田先生にもどります。なんと本年、令和5年6月16日にご逝去されたよし。享年101歳だったそうです。合掌。
山田先生は、1966年に第15代会長を務められたと書かれてました。バリバリの顕微鏡の人だったのね。。。
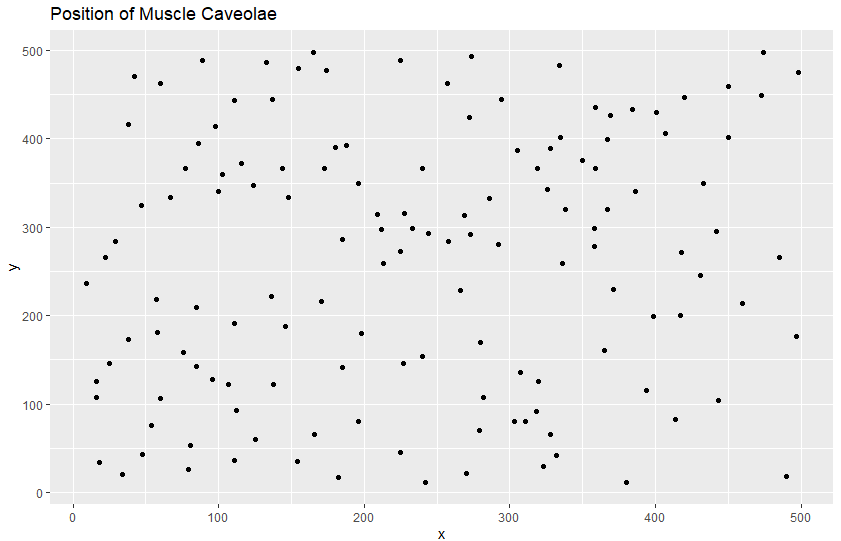
Position of Muscle Caveolae
長々とカベオラについて書いてしまったのは他でもありません。今回のデータセットがあまりにシンプルであるからです。カベオラについてのサンプルデータセットといいつつも、そこに記録されているのはX,Y座標のみです。XY500ユニットの範囲内の位置。筋繊維の2.65μm単位のグリッドだというのですが、2.65、半端。インチ単位なら2.54だろ~。なんなん?
生データとそのプロット
例によって事前にbootパッケージとggplot2をロードしておきます。
library(boot) library(ggplot2)
生データをロードしてみるとこんな感じ。フツーのデータフレームにただx, y座標が記録されているのみ、とくに大きな偏りもないみたい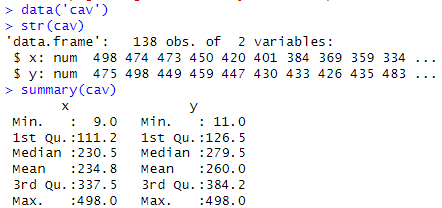 。
。
しかたがないのでXY座標でプロットしてみました。処理はこんなです。
g0 <- ggplot(cav, aes(x=x, y=y))+geom_point()
g1 <- g0+ggtitle("Position of Muscle Caveolae")
g1
素人には一様、かつランダムっぽく、パラパラと存在しているように見えます。このデータをどう処理せよと?密度くらいは計算できるか?やらんけど。