今回もまた未知の世界デス。突然 cd4 と言われてもな。ヘルパT細胞の表面に発現する糖タンパクらしいです。それが何よというと、HIVウイルスの攻撃目標になるみたいです。結果AIDSだと。今回データはそれに対抗するお薬の効き具合のデータらしいのですが、bootstrap法をnestedで適用したらしいっす。何だかな~。
※「データのお砂場」投稿順Indexはこちら
※ R言語向けのパッケージ boot には、サンプルデータセット多数が含まれております。それを ABC順に「舐めて」ます。今回のサンプルデータセットはcd4およびcd4.nestedの2つです。cd4が元データで、cd4.nestedが、元データに対してbootstrap法をnestedで適用した?データらしいっす。
CD4 (Cluster of Differentiation 4)
素人があれこれ書いても受け売りにしかならんので、『日本がん免疫学会』様の以下のページなどご覧くだされや。
それにしてもヘルパーT細胞、時々聞くお言葉だけれど、Tって何よ?という疑問があり。『東邦大学生物分子科学科』様の以下のページで疑問氷解。
素人の勝手な理解では、HIVウイルスはCD4を目印に攻撃をかけてくるので、人体の免疫の司令塔たるヘルパーT細胞が損耗、その結果免疫が落ち、通常なら発症しないような感染症が現れて重篤になるらしいです。よってCD4の損耗具合を測定すればHIVの暴れっぷりが分かるみたい。
cd4 サンプルデータ
今回のデータセットのうち生データにあたるのが、 cd4 です。
CD4 Counts for HIV-Positive Patients
お薬の実験に参加する前と1年後とで2回測定したCD4のカウントデータらしいです。カウントといって100単位ね、と書かれてますけど、単位xxあたりというのはなんだか分かりません。統計のお勉強には不要ってことかい?
もう一つのサンプルデータセットが cd4.nested です。
Nested Bootstrap of cd4 data
上記の cd4 データセットに対して、Bootstrap法、それもNextedなやつを適用した結果らしいです。もう処理してくれているのね。。。
それにしてもねえ。Bootstrap。R言語の boot パッケージは、Bootstrap法のパッケージで、そのお勉強用に多数のサンプルデータが含まれとります。そこそこR言語じゃ「有力」なパッケージだし、Bootstrap法も「統計業界」じゃメジャーみたいです。しかし、BootstrapでWeb検索するとJavaScriptの「フレームワーク」の方ばかりヒット。まあ、統計、と分かるように単語を追加すれば所望のページが見つかりはじめますが。。。しかし、2大巨頭のお陰で、マイコンのRESET後の初期化コード(多分アセンブラ)のBootstrapとか、MOSトランジスタを使った回路の高速化テクであるBootstrapとか、マイナー、しかしこの老人には親しみのあるBootstrapな奴らは遥かな地獄の底に封印されとります。たまには召喚してやりたいっす。つまらぬことを書きました。
Rをつかった統計のブートストラップ法については、『ねこすたっと』様の以下のページがとっても分かりやすかったデス。
『ブートストラップ(bootstrap)法で信頼区間を求める(bootパッケージ)[R]』
先ずは生データ
シンプルなデータフレームです。summaryみるとbaselineの平均は3.288で、oneyearの平均は4.093で、「お薬効いてる感じ」がしないでもないです。
cd4をプロットしてみる
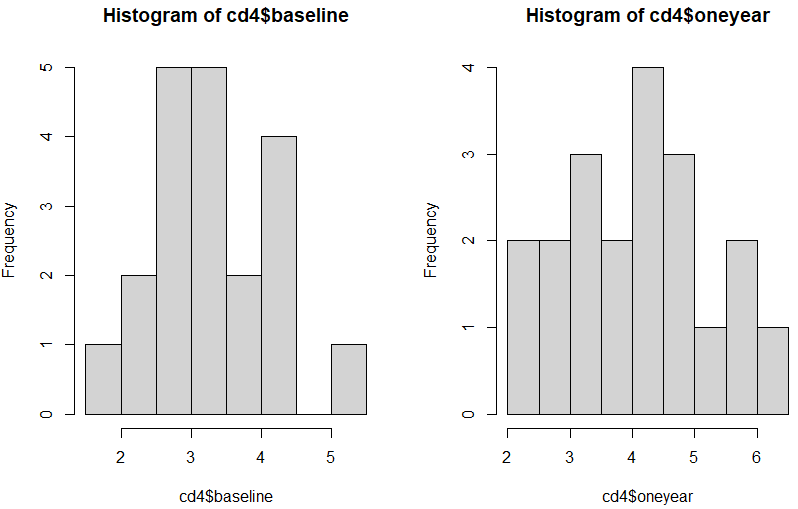
ブートストラップの定石?として、元データのヒストグラムを描いてみます。処理はこんな手順。
layout(matrix(1:2, ncol=2)) hist(cd4$baseline) hist(cd4$oneyear)
上をみると1年後の方が分布が右にズレてる気がしないでもない。
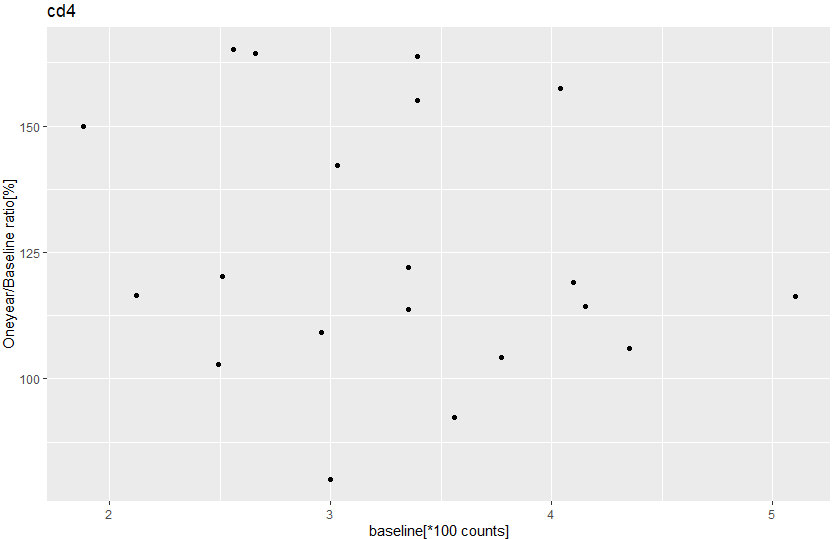
もしや、ということで勝手仮説。元の(baseline)の高い人の方が効果が高いのではと妄想してグラフを描いてみました。こんな感じ。
cd4.ratio <- data.frame(cd4$baseline, cd4$oneyear, (cd4$oneyear/cd4$baseline)*100)
colnames(cd4.ratio) <- c("baseline", "oneyear", "OBratio")
g0 <- ggplot(cd4.ratio, aes(x=baseline, y=OBratio)) + geom_point()
g1 <- g0 + labs(title="cd4", x="baseline[*100 counts]", y="Oneyear/Baseline ratio[%]")
plot(g1)
どうも勝手な妄想だったみたい。引っ込めます。
cd4.nested
Nested Bootstrap法を適用した結果が格納されとります。データをロードしてみるとこんな感じ。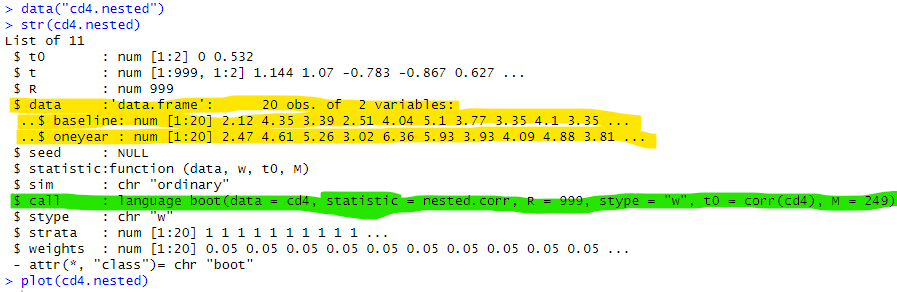
黄色のマーカのところには、元のcd4データそのものが含まれておりますな。そして、緑色のマーカのところには、処理方法までバッチリ書かれてます。しかし、
nested.corr
って何?また調べんと分からんなあ。とりあえず処理結果が以下に。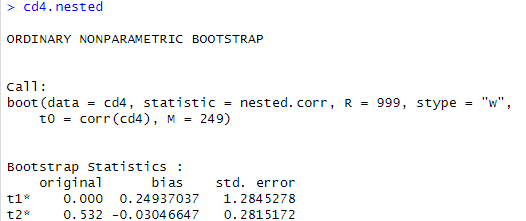
分かる気がしない。。。