Rのパッケージ「Boot」に含まれるサンプルデータセットをabc順に経めぐってます。今回はnodalとな。前立腺ガン関係のサンプルデータセットみたいです。例によって40年前以上の古いデータセットみたいですが、年齢的に他人事とは言えないので怖いデータセットです。ただ0と1が並んでいるだけなんだけれども。
※「データのお砂場」投稿順Indexはこちら
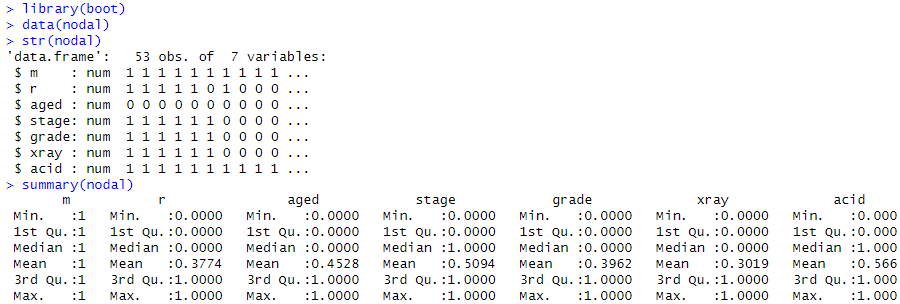
nodal、サンプルデータセット
サンプルデータセットの解説ページが以下に。
Nodal Involvement in Prostate Cancer
上記解説ページによると、前立腺ガンに対する対処方法は、周囲のリンパ節に転移しているか否かが大きく関係しているらしいです(40年以上も前のデータセットの話でです。)穴開けて生体サンプルを取って顕微鏡で見ればハッキリするけれども、そうせずに済ませるような他の方法での診断結果からリンパへの転移を推定するための取得データみたいです。合計7列あるデータですが
-
- m、ただ1が並んでいるだけの列(処理の都合?)
- r、リンパに”involvement”しているか否か(していたら1)
- aged、60歳以上か否か(60歳以上で1)
- stage、触診(肛門経由みたい)でのシビアさ具合の判定(シビアなら1)
- grade、生検結果によるシビアさ具合の判定(シビアなら1)
- xray、レントゲン結果によるシビアさ具合の判定(シビアなら1)
- acid、酸性フォスファターゼのレベル判定(1か0、スレッショルド言及なし)
ということで、ただただ0か1が並んでいるだけのデータセットです。
なお、『岡山大学』様の以下ページから一か所引用させていただくと
前立腺性酸性フォスファターゼ, PAP (prostatic acid phosphatase)
PAPは夜間に低値となる日内変動があり、特に前立腺癌ではその幅が大きく、さらに日差変動も観察されているので、1回のみの測定では不十分で、信頼できる結果を得るには複数回の測定が必要である。
ということであります。また、他のページを拝見すると最近はPAPよりも感度などが良いマーカが使われているみたいです。知らんけど。
まずは生データ
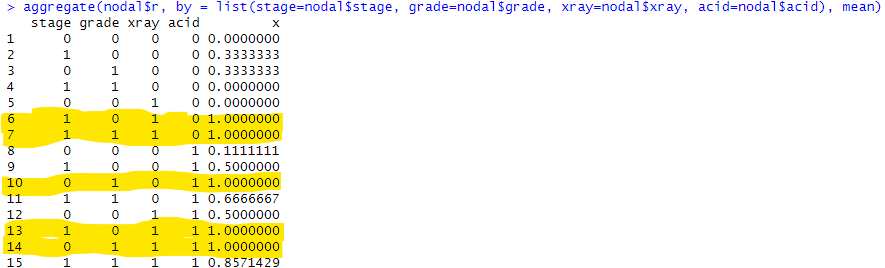
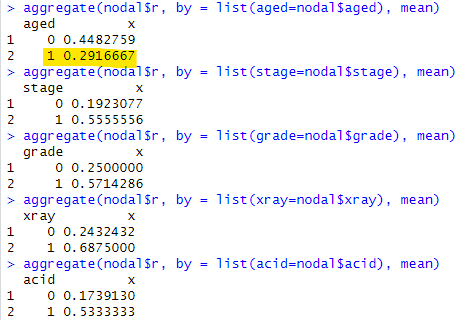
m列は単にデータ列数を集計するための列?なのかね。勝手な解釈でいくと、r列が1の場合がヤバイよという感じか。。。残りの5列、aged、 stage、 grade、 xray、 acidについてそのヤバさ加減をそれぞれの「rの平均値」とってくらべてみました。操作はこんな感じ。
aggregate(nodal$r, by = list(aged=nodal$aged), mean) aggregate(nodal$r, by = list(stage=nodal$stage), mean) aggregate(nodal$r, by = list(grade=nodal$grade), mean) aggregate(nodal$r, by = list(xray=nodal$xray), mean) aggregate(nodal$r, by = list(acid=nodal$acid), mean)
stage、grade、xray、acidについては1判定の場合の「ヤバさ」が0判定より確実に上がってますが、黄色のマーカのようにageでは逆転。年寄より若い人の方がヤバイケースが多いのか。母集団は全て患者様なので。。。
複数の要因の組み合わせ
上記の結果から、ageを除きstage、grade、xray、acidについての判定結果各1をつかって0から15までの16通りに分類したものの「ヤバさ(r)」を計算してみました。使うまいと思っていたのだけれどもFor文使ってしまったのが以下に。元のデータを壊さぬようコピーした上で、列mに結果を代入しちまってます。
nodalw <- nodal
for (i in 1:nrow(nodalw)) {
nodalw[i, 1]<-sum(nodalw[i, 4:7]);
}
aggregate(nodalw$r, by = list(m=nodalw$m), mean)
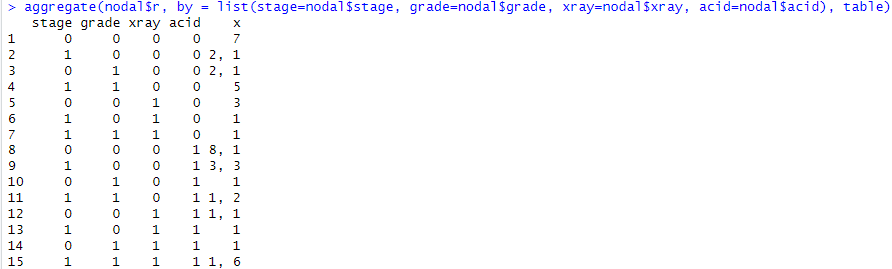
黄色のところが 100% ヤバいケースです。でもね、良く考えるとそれぞれのケースの症例数に大分差がありそう。以下のようにするとrの値毎の件数を数えてくれるみたい。
aggregate(nodal$r, by = list(stage=nodal$stage, grade=nodal$grade, xray=nodal$xray, acid=nodal$acid), table)
数字が2つ書いてあるところは、rが0の時と1の時の集計なのだけれども数字が一つしか書いてない行はどちらか一方です。例えば4行目は5と書いてあるのは全員r=0の5名様で、5行目に3と書いてあるのは全員r=3の3名様です。天と地の違いよな。
まあ、ケース数が少ないのでブートストラップ法で、信頼区間など推定するとよろし、ということなんだろうが、今回も見送り。見送ってばかりじゃないか、自分。